Chọn lọc Single-Nucleotide Polymorphism (SNP) tiềm năng liên quan đến tính kháng Tilapia Lake Virus (TiLV) ở cá rô phi đỏ (Oreochromis spp.)
- Thứ sáu - 26/04/2024 09:56
- In ra
- Đóng cửa sổ này
Cá rô phi đỏ (Oreochromis spp.) là đối tượng nuôi thích hợp và hiệu quả cho vùng đất nông nghiệp năng suất thấp. Đồng bằng sông Cửu Long là khu vực sản xuất cá rô phi...
Cá rô phi đỏ (Oreochromis spp.) là đối tượng nuôi thích hợp và hiệu quả cho vùng đất nông nghiệp năng suất thấp. Đồng bằng sông Cửu Long là khu vực sản xuất cá rô phi chính của cả nước. Trong các bệnh phổ biến trên cá rô phi, Tilapia Lake Virus (TiLV) được báo cáo là mối đe dọa đối với nuôi cá rô phi ở 16 quốc gia (bao gồm Việt Nam) từ bốn châu lục với các đợt bùng phát gây ra tỷ lệ tử vong lên đến 90%. Chính vì vậy, việc chọn lọc con giống cá rô phi đỏ có khả năng kháng TiLV là điều cần thiết.
Trong vài năm qua, RNA-seq đã được ứng dụng trong nhiều nghiên cứu liên quan đến hệ gen chức năng của các loài cá khác nhau, không chỉ ở cá ngựa vằn mà còn ở các loài cá có giá trị thương mại như cá nheo Mỹ (Ictalurus punctatus), cá mú châu Âu (Dicentrarchuslabrax), cá hồi vân (Oncorhynchus mykiss), … Công nghệ RNA-seq đã được dùng để xác định chỉ thị phân tử single nucleotide polymorphism (SNP).
SNP là một thay đổi base đơn lẻ tại một vị trí nhất định trên bộ gen giữa các sinh vật cùng loài. SNP phân bố rải rác khắp bộ gen và được dung phổ biến như là chỉ thị phân tử trong các nghiên cứu về di truyền học và hệ gen. Với độ nhạy cao và độ phân giải ở mức độ một base, RNA-seq được chứng minh là một công cụ rất hiệu quả để nhận diện các SNP ở phạm vi toàn bộ gen. Vì 90% sự khác biệt di truyền giữa các cá thể được ghi nhận bằng các SNP nên SNP là các chỉ thị phân tử vô giá giúp chọn lọc các tính trạng quan trọng trong chọn giống.
Nhóm nghiên cứu của TS. Ngô Huỳnh Phương Thảo, Trưởng phòng Công nghệ sinh học Thủy sản, Trung tâm Công nghệ sinh học thành phố Hồ Chí Minh đã tiến hành công độc cá rô phi (kích thước 5-10 g/con, mật độ là 30 con/bể) với dịch nổi của tế bào E-11 được gây nhiễm với dịch nuôi cấy TiLV (chủng HB196-VN-2020) bằng phương pháp tiêm. Sau công độc, cá có dấu hiệu bệnh lý lâm sàng của việc nhiễm TiLV (chán ăn, sẫm màu, bơi lờ đờ, xung huyết,…) được thu. Nhóm nghiên cứu thu mẫu từ 04 cơ quan (gan, thận ,lách và ruột) của 03 mẫu cá chết đầu tiên và 03 mẫu cá sống sót sau công độc để tách RNA tổng, sau đó gửi giải trình tự RNA bằng hệ thống Illumina. Sàng lọc các SNP tiềm năng bằng các phầm mềm sinh tin học như HISAT2, Galaxy Australia v23.0.,…
Bảng 1. Các SNPs hiện diện tại các mẫu mô cá rô phi đỏ khi so với bộ gen tham chiếu Orenil1.0 của cá rô phi vằn
Trong vài năm qua, RNA-seq đã được ứng dụng trong nhiều nghiên cứu liên quan đến hệ gen chức năng của các loài cá khác nhau, không chỉ ở cá ngựa vằn mà còn ở các loài cá có giá trị thương mại như cá nheo Mỹ (Ictalurus punctatus), cá mú châu Âu (Dicentrarchuslabrax), cá hồi vân (Oncorhynchus mykiss), … Công nghệ RNA-seq đã được dùng để xác định chỉ thị phân tử single nucleotide polymorphism (SNP).
SNP là một thay đổi base đơn lẻ tại một vị trí nhất định trên bộ gen giữa các sinh vật cùng loài. SNP phân bố rải rác khắp bộ gen và được dung phổ biến như là chỉ thị phân tử trong các nghiên cứu về di truyền học và hệ gen. Với độ nhạy cao và độ phân giải ở mức độ một base, RNA-seq được chứng minh là một công cụ rất hiệu quả để nhận diện các SNP ở phạm vi toàn bộ gen. Vì 90% sự khác biệt di truyền giữa các cá thể được ghi nhận bằng các SNP nên SNP là các chỉ thị phân tử vô giá giúp chọn lọc các tính trạng quan trọng trong chọn giống.
Nhóm nghiên cứu của TS. Ngô Huỳnh Phương Thảo, Trưởng phòng Công nghệ sinh học Thủy sản, Trung tâm Công nghệ sinh học thành phố Hồ Chí Minh đã tiến hành công độc cá rô phi (kích thước 5-10 g/con, mật độ là 30 con/bể) với dịch nổi của tế bào E-11 được gây nhiễm với dịch nuôi cấy TiLV (chủng HB196-VN-2020) bằng phương pháp tiêm. Sau công độc, cá có dấu hiệu bệnh lý lâm sàng của việc nhiễm TiLV (chán ăn, sẫm màu, bơi lờ đờ, xung huyết,…) được thu. Nhóm nghiên cứu thu mẫu từ 04 cơ quan (gan, thận ,lách và ruột) của 03 mẫu cá chết đầu tiên và 03 mẫu cá sống sót sau công độc để tách RNA tổng, sau đó gửi giải trình tự RNA bằng hệ thống Illumina. Sàng lọc các SNP tiềm năng bằng các phầm mềm sinh tin học như HISAT2, Galaxy Australia v23.0.,…

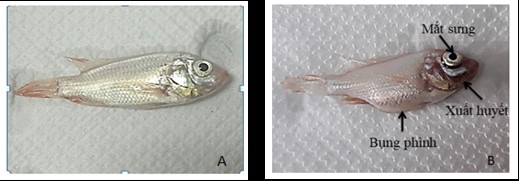
Hình 1. Biểu hiện bệnh lý của cá đối chứng (A) so với cá sau khi tiêm TiLV (B) trong quá trình công độc với chủng HB196-VN-2020
Kết quả cho thấy ở mô gan có 1.460 SNP chỉ hiện diện ở nhóm cá sống và không có mặt ở nhóm cá chết được lọc ra từ dữ liệu SNP của 6 mẫu cá. Số liệu lần lượt cho các mô thận, lách và ruột là 1.939, 6.217 và 896.Bảng 1. Các SNPs hiện diện tại các mẫu mô cá rô phi đỏ khi so với bộ gen tham chiếu Orenil1.0 của cá rô phi vằn
| Loại cá | Số lượng SNP so với bộ gen tham chiếu Orenil1.0 |
Số lượng SNP chỉ hiện diện ở nhóm cá sống sau quá trình công độc TiLV | ||||||
| Gan | Thận | Lách | Ruột | Gan | Thận | Lách | Ruột | |
| Cá sống 1 | 33.130 | 69.409 | 54.984 | 47.172 | 1.460 | 1.939 | 6.217 | 896 |
| Cá sống 2 | 35.889 | 94.746 | 73.004 | 61.618 | ||||
| Cá sống 3 | 61.125 | 74.428 | 103.301 | 28.980 | ||||
| Cá chết 1 | 30.131 | 71.628 | 2.820 | 32.228 | - | - | - | - |
| Cá chết 2 | 33.965 | 62.364 | 34.031 | 55.254 | ||||
| Cá chết 3 | 31.629 | 80.188 | 65.358 | 4.273 | ||||
Gan, thận, ruột và lách đều là những cơ quan quan trọng, liên quan đến con đường xâm nhiễm của TiLV và khả năng tạo ra kháng thể của cá rô phi đỏ. Đây là nghiên cứu đầu tiên ở Việt Nam về hệ gen phiên mã của cá rô phi đỏ sống và chết sau khi công độc với chủng TiLV. Tổng cộng 10.512 SNP tiềm năng được tím thấy từ dữ liệu hệ gen phiên mã của bốn mẫu mô (gan, thận, lách và ruột) có thể được ứng dụng để phát triển chip SNP nhằm chọn giống cá rô phi đỏ có sức đề kháng lại sự xâm nhiễm của TiLV. Kết quả của nghiên cứu hiện tại còn mang tính bước đầu nên cần phải được tím hiểu sâu hơn về mối liên hệ của các SNP này với tính kháng TiLV của cá rô phi đỏ với số lượng mẫu lớn hơn.
Nghiên cứu này đã được công bố trên tạp chí Nông nghiệp và Phát triển nông thôn số tháng 10/2023.
Nghiên cứu này đã được công bố trên tạp chí Nông nghiệp và Phát triển nông thôn số tháng 10/2023.